Pesquisa Grupo Carla Polycarpo
tRNAs! Já pensou em quão interessantes são essas moléculas?
As informações contidas nos códons dos RNAs mensageiros (mRNA) são decifradas pelos anticódons presentes nos RNAs de transferência (tRNAs). Os níveis individuais de tRNAs e a cinética do pareamento códon-anticódon determinam as taxas de tradução de códons sinônimos (isto é, códons que codificam o mesmo aminoácido). Evidências mostram que mudanças na composição de tRNAs são parte de um programa mais fino da regulação da tradução, com perfis de expressão que podem variar dependendo da situação fisiológica da célula, e que estão em coordenação com as mudanças na expressão gênica.
As mudanças na expressão gênica são a base de processos fisiológicos cruciais como proliferação, diferenciação e apoptose, enquanto a regulação aberrante da expressão gênica leva a doenças, incluindo câncer e doenças neurodegenerativas.
Embora a eficácia da tradução seja determinada em parte pelo repertório de tRNAs, pouco se sabe sobre como as mudanças dos níveis de diferentes isoaceptores de tRNAs, assim como mudanças nas moléculas de tRNAs e enzimas correlacionadas, afetam as funções celulares.
A síntese de polipeptídeos, só é possível devido à presença de duas regiões principais nas moléculas de tRNA, a região 3’-CCAOH, onde se liga o aminoácido, e a região do anticódon, que se pareia ao códon no mRNA e determina a identidade do aminoácido a ser inserido na cadeia polipeptídica. A perfeita função do tRNA exige a mauração prévia dessas moléculas seguida da adição do aminoácido correspondente pelas chamadas aminoacil-tRNA sintetases. Estas enzimas adicionam os aminoácidos correspondentes ao anticódon na região 3’CCAOH do tRNA, que portanto, representa um requisito fundamental para todo o processo.
Assim como acontece com a grande maioria das moléculas sintetizadas pelas células, muitos RNAs, após serem transcritos, precisam sofrer um processo de maturação, que inclui adição ou remoção de sequências e edição de nucleotídeos. A biossíntese de tRNAs maduros e funcionais é um processo extremamente complexo que envolve uma série de passos e reações enzimáticas. Entre eles podemos citar a (i) remoção da região terminal 5’ líder pela RNAase P; (ii) remoção da extensão na região 3’; (iii) adição do grupamento -CCAOH à região terminal 3’ em eucariotos, algumas eubactérias e algumas archaeas; (iv) remoção dos íntrons por uma endonuclease e a junção das duas metades resultantes por uma ligase; (v) as próprias modificações pós-transcricionais em certas bases do tRNA.
As modificações pós-transcricionais são encontradas em todos os domínios da vida. Todas as espécies de tRNAs já caracterizadas apresentam numerosas modificações, seja nas bases nitrogenadas ou na ribose formadora da estrutura dos nucleotídeos.A maioria dos nucleosídeos modificados parece aumentar a performance de tRNAs maduros, fazendo com que sejam mais eficientes, aumentando acurácia na síntese de proteínas.
Em nosso grupo, usando como modelos biológicos insetos hematófogos, tripanossomatídeos e células humanas, procuramos entender o processo de maturação dos tRNAs, o papel da modificação de suas bases na regulação da expressão gênica, como situações de estresse podem levar à aminoacilação errônea do tRNA, levando à incorporação de aminoácidos errados em posições específicas em proteínas (o que pode levar à disfunção destas proteínas) e o repertório de tRNAs em diferentes situações fisiológicas e como isso pode afetar a expressão gênica.
Maturação de tRNAs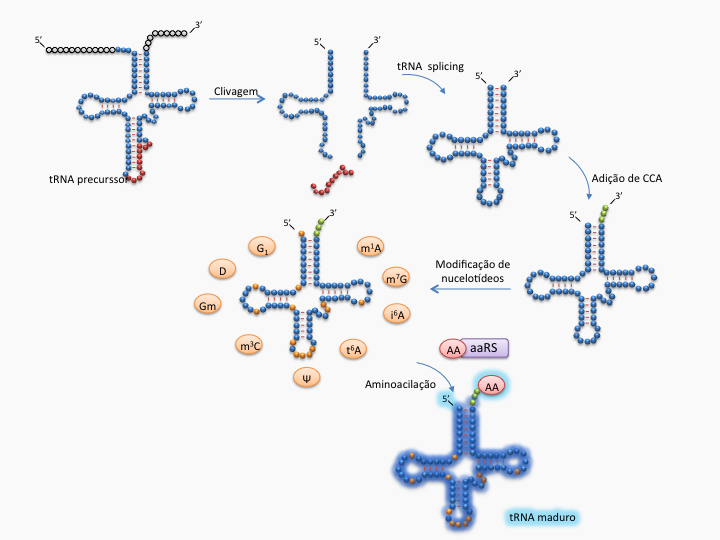
 Possui graduação em Microbiologia e Imunologia pela Universidade Federal do Rio de Janeiro (1997), mestrado em Ciências (Microbiologia) pela Universidade Federal do Rio de Janeiro (1999) e doutorado em Ciências (Microbiologia) pela Universidade Federal do Rio de Janeiro (2003) com doutorado sanduíche e Pós-doc na Universidade de Yale (2006) no Departamento de Biofísica Molecular e Bioquímica. Atualmente é Professor Adjunto da Universidade Federal do Rio de Janeiro. Trabalhou durante ...
Possui graduação em Microbiologia e Imunologia pela Universidade Federal do Rio de Janeiro (1997), mestrado em Ciências (Microbiologia) pela Universidade Federal do Rio de Janeiro (1999) e doutorado em Ciências (Microbiologia) pela Universidade Federal do Rio de Janeiro (2003) com doutorado sanduíche e Pós-doc na Universidade de Yale (2006) no Departamento de Biofísica Molecular e Bioquímica. Atualmente é Professor Adjunto da Universidade Federal do Rio de Janeiro. Trabalhou durante ...